Usługa sekwencjonowania wykorzystywana jest w coraz większej ilości różnych dziedzin życia. Od wielu lat z pomocą w przeprowadzeniu analizy DNA przychodzą zautomatyzowane sekwenatory. Jednak jak dokładnie przebiega ten proces zza kulis? W tym artykule postaramy się przybliżyć techniczne aspekty usługi sekwencjonowania w technologii długich odczytów DNA, czyli technologii nanoporowej.
Sekwencjonowanie nanoporowe w praktyce
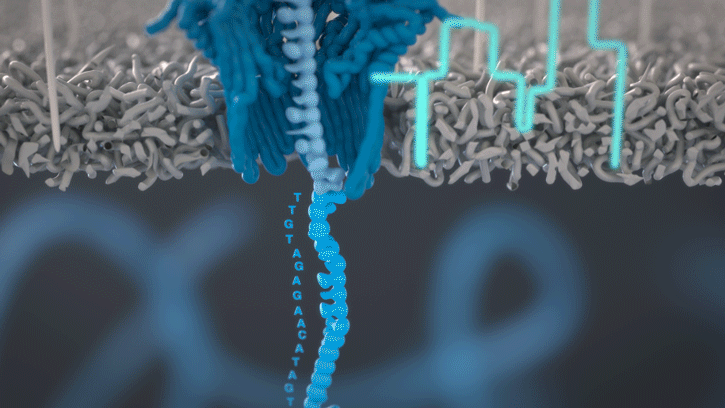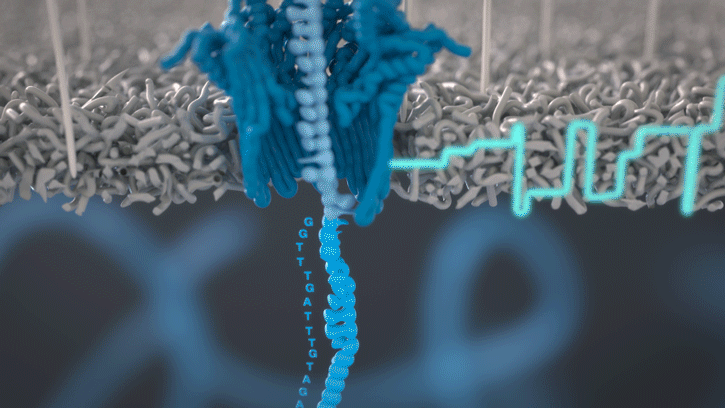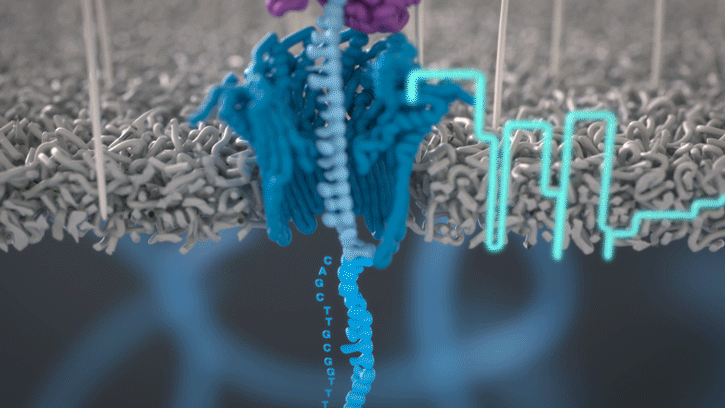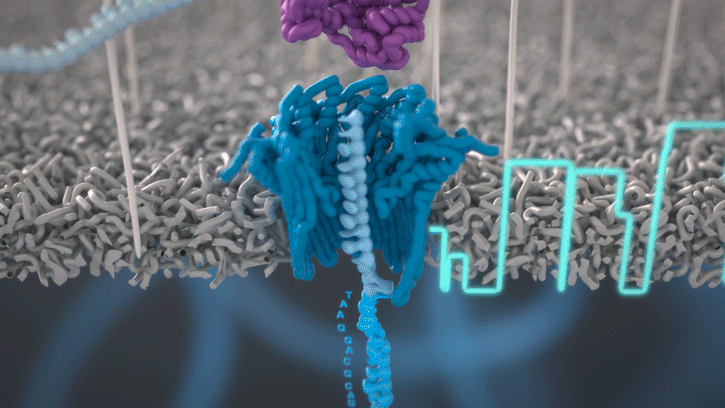
Pory to białkowe struktury umożliwiające przepływ jonów przez błonę biologiczną. Niektóre z nich tworzą kanały o średnicy umożliwiającej translokacje nie tylko jonów, ale także dużych makromolekuł. Wśród porów posiadających powyższe właściwości można wyróżnić m.in. alfa-hemolizyny, mspA – poryny produkowane przez mykobakterie czy białka csgG, tworzące kanały sekrecyjne u E. coli.
Pory wykorzystywane w procesie sekwencjonowania, określane są mianem nanoporów. To genetycznie zmodyfikowane białka, które uprzednio zostały wyselekcjonowane pod kątem efektywności translokacji kwasów nukleinowych i rozmieszczone w syntetycznej membranie. Przejściu kwasu nukleinowego z jednej strony błony na drugą towarzyszy zmiana potencjału elektrycznego, charakterystyczna dla konkretnej sekwencji nukleotydowej. O tym, w jaki sposób sygnał elektryczny zamieniany jest na sekwencję nukleotydową napisaliśmy w publikacji poświęconej tematowi basecalling. Aby nić DNA lub RNA mogła w sposób kontrolowany przemieszczać się wzdłuż nanoporu konieczne jest dołączenie do niej cząsteczki białkowej, zwanej adaptorem. Adaptor białkowy pełni dwie ważne funkcje: rozplata helisę, jak i nadaje ściśle określoną prędkość przechodzenia cząsteczek kwasów nukleinowych przez por.
Technologia długich odczytów DNA
Nanopore, jako platforma oferuje odczyty o N50 >10 kb, przez co zdecydowanie przewyższa technologie sekwencjonowania II generacji. Tak naprawdę długość odczytu jest ograniczona jedynie długością analizowanego fragmentu DNA. Długie odczyty posiadają szeroki zakres zastosowań w genomice organizmów modelowych i niemodelowych, jak również w diagnostyce klinicznej. Przede wszystkim podwyższają prawdopodobieństwo prawidłowego mapowania, dzięki czemu umożliwiają składanie genomów de novo (de novo assembly), czyli bez wcześniejszej znajomości sekwencji referencyjnej dla danego organizmu. Ponadto analiza dziesiątek tysięcy nukleotydów w pojedynczym odczycie daje unikatową możliwość wykonania analizy strukturalnych aberracji chromosomowych, umożliwiając tym samym wykrycie i analizę rozległych mutacji strukturalnych takich jak delecje, insercje, inwersje czy translokacje. Kolejną przewagą technologii III generacji jest możliwość analizy dużych regionów powtórzonych, niedostępną dla sekwencjonowań opartych o generowanie krótkich odczytów (<1000 pz). Detekcja i dokładna kwantyfikacja zmiennej liczby elementów powtórzonych jest szczególnie przydatna w diagnostyce takich chorób jak zespół łamliwego chromosomu X czy pląsawicy Huntingtona. Dodatkowo umożliwia korelowanie funkcjonalności białka z ilością powtórzonych kopii kodującego je genu, jak w przypadku AMY1. Nanopory dają także możliwość sekwencjonowania natywnych kwasów nukleinowych, dzięki czemu eliminuje się błędy związane z amplifikacją, które mogą wynikać na przykład z preferencyjnego wiązania starterów do niektórych matryc DNA. Brak konieczności wykonania reakcji PCR skutkuje również możliwością analizy poziomu metylacji sekwencjonowanych nici.